Das Robert-Koch-Institut (RKI) stellt die kumulativen Corona-Fallzahlen in einer Excel-Tabelle bereit. Sonderlich schön ist die Tabelle nicht, aber sie liefert die Daten.
Auf dem Blatt „Fälle-Todesfälle-gesamt“ sind die Fälle und Todesfälle nach Tagen in Zeilen aufgeführt. Die anderen Blätter enthalten Daten der Bundesländer, die hier nicht interessieren.
Für eigene Auswertungen möchte ich die Tabelle herunterladen und in eine SQL-Datenbank (MariaDB) importieren. Die anschließende Auswertung erfolgt mittels R Script.
Herunterladen der Tabelle
Wenn ich versuche, die Datei mit wget herunter zu laden, gibt es den Fehler 403. Also laden wir dei Datei mit curl herunter. Anschließen wird das erste Spreadsheet mit ssconvert aus dem Paket Gnumetrics in eine CSV-Datei konvertiert. Leider funktioniert ssconvert nicht als Filter.
Konvertieren der Daten
Anschließend wird mit sed und awk einen SQL-Batch Job erstellt, der die Datei per INSERT in die Tabelle einfügt.
Warum nicht mittels IMPORT?
- Der Zugriff auf ein File ist durch den SQL Server ist nicht immer möglich.
- Die exportierte CSV-Datei ist grottenschlecht formatiert.
- Die Tabelle enthält am Ende Zeilen ohne Daten.
Hier vier Beispielzeilen:
"25.08,2020",234853,,1278,9277,5,"0,03950130507168314",225576
01.09.2020,243599,,1218,9302,4,"0,038185706837877004",234297
2020/10/05,300619,,1382,9534,5,"0,03171456228648222",291085
2020/10/06,,,,,,,
Ob dies an der Excel Tabelle oder ssconvert liegt, habe ich nicht geprüft.
Da es nur um die Zahlen der Fälle und Todesfälle geht, wird der Schrott von mittels sed gelöscht. Das Datum in der RKI Tabelle ist immer 0 Uhr des Folgetages, nicht 24 Uhr des Tages an dem die Fälle aufgetreten sind. Dies wird beim Import „korrigiert“.
Importieren der Daten
Im letzten Schritt können wir jetzt die Daten in die Datenbank importieren.
Folgendes Script führt diese Aufgaben aus.
#!/bin/bash URL="https://www.rki.de/DE/Content/InfAZ/N/Neuartiges_Coronavirus/Daten/Fallzahlen_Kum_Tab.xlsx?__blob=publicationFile" #TEMP=$(mktemp -d) TEMP="/tmp/rki" DEST="$TEMP/rki.xlsx" CSV="$TEMP/rki.csv" SQL="$TEMP/rki.sql" curl --output "$DEST" --url "$URL" ssconvert --export-type=Gnumeric_stf:stf_csv -O 'sheet=Fälle-Todesfälle-gesamt' "$DEST" "$CSV" ( cat <<EOF USE COVID19; DROP TABLE IF EXISTS rki; CREATE TABLE rki (day DATE primary key, cases BIGINT, deaths BIGINT); INSERT INTO rki VALUES ("2020-02-24",0,0); EOF sed '1,3d; s#"[^"]*",##; s#,,#,0,#g; s#,,#,#g' "$CSV" \ | awk -F ',' 'BEGIN {i=1}; {if ($2>0) print("INSERT INTO rki VALUES ( ADDDATE(\"2020-02-25\"," i ")," $2 "," $5 ");" );i=i+1}' ) > "$SQL" mysql -u rscript -p rscript < "$SQL"
Der Nutzer „rscript“ benötigt Zugriff schreibenden auf die Datenbank COVID19. Das Script und weitere befindet sich auf GitHub unter: R-Example
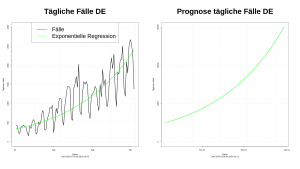
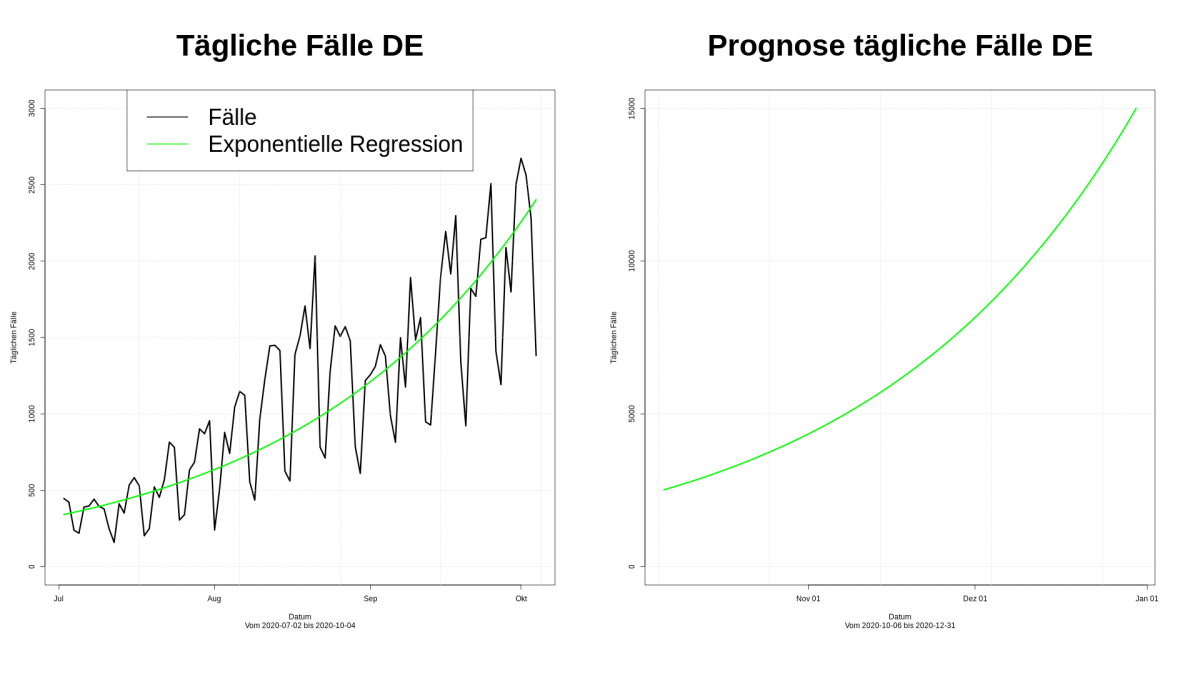
Nun stehen uns die Daten in der Datenbank zur Auswertung bereits. Für die Auswertung und das Zeichnen von Diagrammen bietet sich R an. Doch das ist einen neuen Beitrag wert.
Das folgende Beispiel ergibt die täglichen Fälle:
use COVID19;
select
t1.day,
t1.cases-t2.cases as Cases,
t1.deaths-t2.deaths as deaths
from rki as t1
inner join rki as t2
on t1.day=adddate(t2.day,1);
Neueste Kommentare